El ADN, la molécula de la vida, aparece dentro de los organismos vivos estirado, retorcido y doblado de diversas maneras. Las simulaciones por ordenador han ayudado mucho a esclarecer cómo estas deformaciones influyen en su funcionamiento
Las simulaciones por ordenador son una herramienta indispensable para comprender el funcionamiento de moléculas cruciales para los seres vivos, ya que permiten acceder a detalles microscópicos y ofrecen mucha más información que la obtenida en experimentos. Sin embargo, el resultado de una simulación depende del modelo utilizado, es decir, de cómo describimos las interacciones entre los átomos de la molécula.
Recientemente, en un trabajo publicado en la revista Journal of Chemical Theory and Computation, investigadores de la Universidad Autónoma de Madrid (UAM) han descubierto que, al contrario de lo que se pensaba, la elección del modelo es fundamental.
Para determinar qué modelo se ajusta mejor a la realidad, los investigadores compararon sus resultados con experimentos, específicamente examinando las propiedades elásticas del ADN, que indican el costo de estirar, doblar o retorcer la molécula.
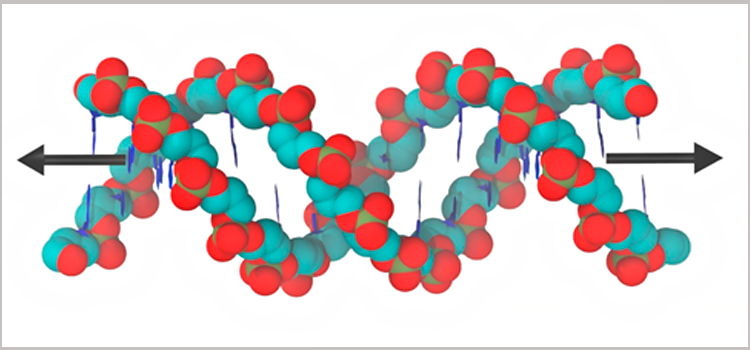
Representación de hebras de ADN. / Roldán-Piñero, C.; Luengo-Márquez, J.; Assenza, S.; Pérez, R.
“La elasticidad del ADN es crucial para decidir qué sección del código genético se va a leer, ya que determina cómo se comprime la doble hélice en el interior de la célula. Por lo tanto, es vital saber qué modelos son más adecuados para describir estas propiedades”, explican los investigadores.
Un nexo común
Aunque los científicos encontraron discrepancias significativas entre los modelos, identificaron un nexo común: al hacer un zoom en la estructura atómica de la doble hélice de ADN, observaron que está "encorvada", es decir, que cada segmento presenta cierta curvatura.
De este modo, los investigadores descubrieron que, independientemente del modelo, es posible predecir la elasticidad de la molécula a partir de su curvatura. “El uso de ordenadores ha revelado una relación entre una propiedad microscópica y la elasticidad del ADN, una característica que afecta a moléculas con tamaños y secuencias muy diferentes, destacando así los elementos comunes en lo aparentemente diverso”, detallan los autores.
En las simulaciones se estiraron moléculas de ADN con distintos códigos genéticos, tratando de reproducir las condiciones de laboratorio. Durante las simulaciones, se tomaron ‘fotografías’ de las posiciones de la molécula a lo largo del tiempo. Comparando las ‘películas’ resultantes, los científicos pudieron comprender la respuesta del ADN a esas deformaciones y calcular sus propiedades elásticas.
Ciertamente, la realización de simulaciones requiere un profundo conocimiento de las leyes que rigen las interacciones entre los átomos del ADN. Aunque estas leyes son de naturaleza cuántica, realizar simulaciones siguiendo esas normas es una tarea inabordable para los ordenadores actuales.
“Por tanto, el papel de estos modelos es imitar las consecuencias de esas interacciones cuánticas de forma que las simulaciones sean viables. Esta descripción átomo a átomo sirve también como base para desarrollar modelos menos detallados que permitan simular moléculas más grandes y complejas”, concluyen los investigadores.
Referencia bibliográfica: Roldán-Piñero, C.; Luengo-Márquez, J.; Assenza, S.; Pérez, R. (2024). Systematic Comparison of Atomistic Force Fields for the Mechanical Properties of Double-Stranded DNA. J Chem Theory Comput. 2024 Mar 12;20(5):2261-2272. doi.org/10.1021/acs.jctc.3c01089

