Este trabajo podría permitir el desarrollo de nuevos tratamientos para minimizar las secuelas ocasionadas por los infartos de miocardio
Investigadores de la Universidad Carlos III de Madrid (UC3M) han desarrollado un sistema basado en técnicas de visión artificial que permite realizar un análisis automático de vídeos biomédicos capturados mediante microscopía para poder caracterizar y describir el comportamiento de las células que aparecen en las imágenes.
Estas nuevas técnicas desarrolladas por el equipo de ingenieros de la UC3M se han utilizado para realizar medidas en tejidos vivos en una investigación realizada con científicos del Centro Nacional de Investigaciones Cardiovasculares (CNIC). Gracias a ello, el equipo descubrió que los neutrófilos (un tipo de células inmunes) muestran diferentes conductas en la sangre durante los procesos inflamatorios y han identificado que una de ellas, provocada por la molécula Fgr, está asociada al desarrollo de enfermedades cardiovasculares. Este trabajo, publicado recientemente en la revista Nature, podría permitir el desarrollo de nuevos tratamientos para minimizar las secuelas ocasionadas por los infartos de miocardio. En el estudio han participado investigadores de la Fundación Vithas, de la Universidad de Castilla La Mancha, la Agencia de Ciencia y Tecnología de Singapur (ASTAR) y la Universidad de Harvard (EEUU), entre otros centros.
“Nuestra contribución consiste en el diseño y desarrollo de un sistema completamente automático, basado en técnicas de visión artificial, que permite caracterizar las células objeto de estudio mediante el análisis de vídeos capturados por los biólogos a través de la técnica de microscopía intravital”, indica uno de los autores de este trabajo, el catedrático Fernando Díaz de María, responsable del Grupo de Procesado Multimedia de la UC3M. Se han realizado medidas automáticas de la forma, tamaño, movimiento y posición respecto al vaso sanguíneo de unos pocos miles de células, en comparación con los estudios biológicos tradicionales que se suelen sustentar en análisis de unos pocos cientos de células caracterizadas manualmente. De esta forma, se ha podido llevar a cabo un análisis biológico más avanzado y con mayor significancia a nivel estadístico.
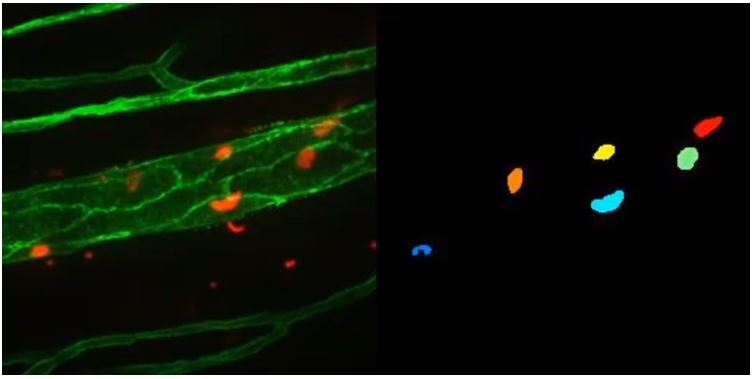
Segmentación de neutrófilos mediante el sistema propuesto con el software ACME. La segmentación es 3D, pero se muestra una versión acumulada en 2D. A la izquierda, imagen original de microscopía: vasos sanguíneos (verde) y neutrófilos (rojo). A la derecha, segmentación automática mediante ACME (un color por cada célula).
Este nuevo sistema tiene diversas ventajas, según los investigadores, en términos de tiempo y precisión. En general, “no resulta viable mantener a un biólogo experto segmentando y siguiendo células en vídeos durante meses. En cambio, por dar una idea aproximada (porque depende de la cantidad de células y profundidad del volumen 3D), nuestro sistema tarda apenas un cuarto de hora en analizar un vídeo de 5 minutos”, señala otro de los investigadores, Ivan González Díaz, profesor titular del Dpto. de Teoría de la Señal y Comunicaciones de la UC3M
Las redes neuronales profundas, las herramientas en las que se apoyan estos ingenieros para la segmentación y detección de las células, son básicamente algoritmos que aprenden a partir de ejemplos, por lo que para desplegar el sistema en un nuevo contexto es necesario generar suficientes ejemplos que permitan su entrenamiento. Estas redes forman parte de las técnicas de aprendizaje automático (machine learning), que a su vez es una disciplina dentro del campo de la Inteligencia Artificial (IA). Además, el sistema incorpora a su vez otro tipo de técnicas estadísticas y modelos geométricos, todos ellos descritos en otro trabajo publicado recientemente en la revista Medical Image Analysis.
El software que implementa el sistema resulta versátil y se puede adaptar a otros problemas en pocas semanas. “De hecho, lo estamos aplicando ya en otros escenarios diferentes, estudiando el comportamiento inmunológico de las células-T y células dendríticas en tejidos cancerosos. Y los resultados provisionales son prometedores”, adelanta otro de los investigadores del equipo de la UC3M, Miguel Molina Moreno.
En cualquier caso, a la hora de investigar en este campo, los investigadores destacan la importancia del trabajo de un equipo interdisciplinar. “En este contexto, es importante valorar el esfuerzo previo de comunicación entre biólogos, matemáticos e ingenieros requerido para entender los conceptos básicos de otras disciplinas antes de poder hacer progresos reales”, concluye Fernando Díaz de María.
Más información:
Crainiciuc, G., Palomino-Segura, M., Molina-Moreno, M., González-Diaz, I., Díaz-de-María, F., Hidalgo, A. Behavioural immune landscapes of inflammation. Nature 601, 415–421 (2022). https://doi.org/10.1038/s41586-021-04263-y
Molina-Moreno, M. González-Díaz, I. Sicilia, J. Crainiciuc, G. Palomino-Segura, M. Hidalgo, A. Díaz-de-María, F. (2022). ACME: Automatic feature extraction for cell migration examination through intravital microscopy imaging. Medical Image Analysis, v. 77, 102358. https://doi.org/10.1016/j.media.2022.102358

