La pandemia de la COVID-19 ha revolucionado los modelos utilizados en epidemiología para predecir la evolución del comportamiento de nuevos patógenos. Hasta ahora se han estado empleando los modelos compartimentales para el estudio matemático de las enfermedades infecciosas, donde la población se divide en compartimentos y en cada uno de ellos los individuos tienen unas características determinadas, es decir, pueden estar en un estado susceptible de infectarse, infectados o recuperados (modelos SIR, por las siglas de “Susceptible-Infected-Recovered”).
Cuando surgió el virus SARS-COV-2, causante de la enfermedad de la COVID-19, se emplearon estos modelos en los que se pronosticaba que, según los contactos de un individuo en la sociedad, un infectado podría infectar a un susceptible. Sin embargo, como se ha observado posteriormente, esto modelos no se ajustaban de forma totalmente satisfactoria a la realidad de la evolución de la pandemia al no tener en cuenta otras variables.
El trabajo realizado por la URJC y la Universidad Bar-Ilan (Israel) ha estudiado por primera vez de manera conjunta dos factores relevantes a la hora de realizar los modelos matemáticos: la velocidad con la que un virus se propaga dentro de una población de individuos y la rapidez a la que puede mutar un patógeno cuando ha infectado a un individuo.
Tal y como apuntan los investigadores en el artículo publicado en la prestigiosa revista científica Nature Communications: “Si las mutaciones son demasiado lentas, la prevalencia del patógeno decae antes de la aparición de una mutación crítica. Por otro lado, si las mutaciones son demasiado rápidas, la evolución del patógeno se vuelve volátil y, una vez más, no se propaga. Sin embargo, entre estos dos extremos, identificamos una amplia gama de condiciones en las que un patógeno inicialmente subpandémico puede avanzar para obtener una prevalencia generalizada”.
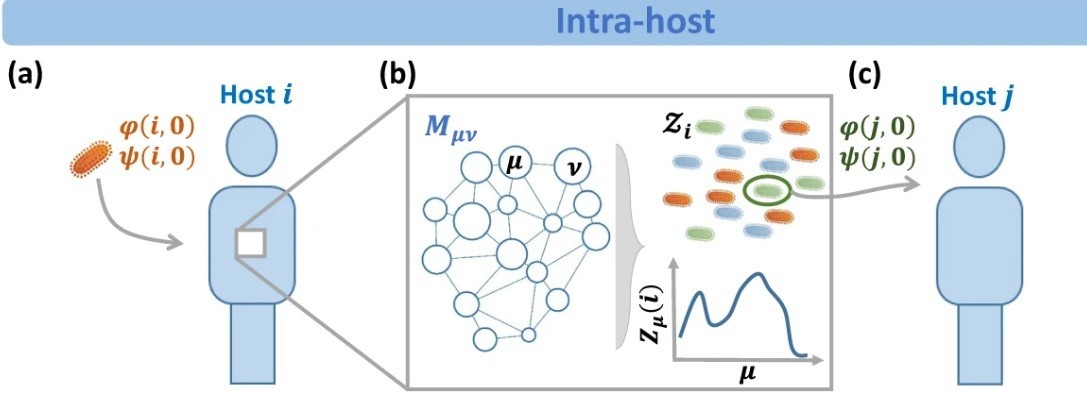
Imagen: Interacción entre la dinámica evolutiva y epidemiológica.
Estas condiciones a las que hacen referencia en este estudio son las que se han conjugado para que el SARS-COV-2 se haya propagado a escala mundial y con tanta rapidez. “Para hacer los modelos epidemiológicos más capaces de capturar la realidad de una pandemia es necesario considerar a la vez dos mecanismos: la propagación y la mutación del patógeno”, subraya Stefano Boccaletti, investigador del Grupo de investigación consolidado en Análisis y Computación con Redes Complejas de la URJC, dirigido por el profesor Regino Criado, y doctor honoris causa en 2014.
El modelo desarrollado por el investigador de la URJC y Baruch Barzel, científico del departamento de Matemáticas de la Universidad Bar-Ilan, servirá para prever futuras pandemias que puedan estar causadas por patógenos que tengan gran capacidad de mutación y propagación en condiciones similares. En el análisis que presentan, utilizan diferentes variables temporales, de replicación del virus o empleo de fármacos que, mediante diferentes ecuaciones matemáticas, arrojan resultados relevantes de cómo evolucionó la pandemia y cómo podrían desarrollarse nuevas enfermedades. “Hasta ahora, todas las fases de mutaciones apenas se habían considerado en el proceso de predicción y este es uno de los primeros trabajos que tiene en cuenta este factor, junto con la velocidad de propagación, de manera explícita”, apunta Stefano Boccaletti.
Para desarrollar este trabajo y recabar los numerosos datos analizados, la URJC y la Universidad Bar-Ilan también han contado con la colaboración de científicos de las universidades chinas de Jinan, Tecnológica de Zhejiang, Jiangsu y Normal del Este de China.
Referencia bibliográfica:
Zhang, X., Ruan, Z., Zheng, M. et al. Epidemic spreading under mutually independent intra- and inter- host pathogen evolution. Nat Commun 13, 6218 (2022). https://doi.org/10.1038/s41467-022-34027-9.

