Este exhaustivo trabajo acelera el conocimiento del papel de la microbiota y la generación de una nueva modalidad de fármacos y nutracéuticos para enfermedades cardiovasculares, el párkinson, el alzhéimer, enfermedades infecciosas o el cáncer
Un reciente análisis computacional desarrollado desde el Instituto IMDEA Alimentación ha permitido identificar una gran cantidad de proteínas humanas y bacterianas que están involucradas en la función de la microbiota de múltiples procesos biológicos para la salud, como los vinculados al sistema inmunitario, el metabolismo, la nutrición o el sistema nervioso. La información obtenida del estudio servirá para diseñar nuevos fármacos capaces de modular su actividad a partir de mecanismos que, hasta el momento, no se han explorado.
El grupo de investigadores de la Unidad de Bioestadística y Bioinformática, liderados por el Dr. Gonzalo Colmenarejo, ha realizado y validado predicciones de hasta 9700 interacciones diferentes entre las moléculas sintetizadas por las bacterias del intestino -los metabolitos microbianos-, y proteínas humanas y bacterianas que ahora se publican, por primera vez y de forma abierta, para acelerar el conocimiento del funcionamiento de la microbiota intestinal y su potencial uso terapéutico. Este hecho se está aprovechando para explotar nuevas dianas terapéuticas que “imitan” estas moléculas, dirigidas a áreas muy diversas como las enfermedades cardiovasculares, el párkinson, el alzhéimer, las enfermedades infecciosas y el cáncer, ya con algunos compuestos en fases clínicas.
En los últimos años, el papel que la microbiota tiene en la salud está cobrando cada vez mayor relevancia. El cuerpo humano alberga billones de células microbianas, especialmente en el intestino, y muchos procesos biológicos están influidos por los sistemas de señalización que se establecen entre las células del huésped y las de la propia microbiota. Esta comunicación bidireccional se ejerce a través de esos metabolitos que se unen a las proteínas, desencadenando así una respuesta biológica.
A partir de este trabajo de IMDEA Alimentación, «hemos podido identificar patrones hasta ahora desconocidos que relacionan las estructuras y clase química de estos compuestos, y la clase y origen biológico de las proteínas», explica el jefe del Grupo, Gonzalo Colmenarejo. En referencia al proceso que se ha llevado a cabo, el Dr. Colmenarejo detalla que se ha empleado el cribado virtual para «ampliar más de 4 veces» el conjunto de las interacciones moleculares, validarlas con análisis retrospectivos y priorizarlas con herramientas bioinformáticas en función de la relevancia biológica, y de esa forma, «llenar los vacíos que existían en todo lo publicado hasta ahora», recalca.
Pruebas experimentales rápidas y enfocadas
Además de permitir el diseño racional de nuevas modalidades de fármacos, el uso de estos metabolitos microbianos es la forma de ampliar el espacio de dianas terapéuticas en aproximadamente dos órdenes de magnitud, el tamaño del vasto genoma microbiano. El análisis ha identificado 9711 interacciones, que involucran a 752 metabolitos, los cuales son capaces de unirse a 180 proteínas microbianas y 1047 humanas.
En concreto, se han encontrado interacciones completamente nuevas en 125 combinaciones de clase química vs familia de proteína desconocidas hasta el momento, e incrementado en otras 326 combinaciones. Este trabajo se ha publicado en el Journal of Chemical Information and Modeling, prestigiosa revista de la American Chemical Society, y en él también han participado los investigadores Cristian Orgaz y Andrés Sánchez-Ruiz.
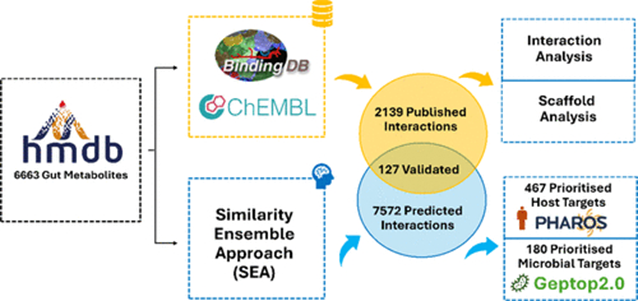
Esquema que describe el flujo de trabajo para los análisis y las herramientas utilizadas en este trabajo.
Las miles de interacciones encontradas están ya a disposición de investigadores de todo el mundo para que puedan ser probadas de forma rápida y con gran probabilidad de éxito. «La alternativa sería probar de forma ciega todos los metabolitos con todas las proteínas, un esfuerzo que requeriría gran cantidad de tiempo y recursos», apunta Gonzalo Colmenarejo, quien aboga por la investigación abierta «porque desde los centros públicos podemos generar un conocimiento fundamental que, al ser compartido, supone un salto cualitativo de cara al descubrimiento de fármacos en muchas áreas terapéuticas», en este caso para la prevención y tratamiento de enfermedades actuando sobre la microbiota intestinal.
Referencia bibliográfica: Cristian Orgaz, Andrés Sánchez-Ruiz & Gonzalo Colmenarejo. Identifying and Filling the Chemobiological Gaps of Gut Microbial Metabolites. Journal of Chemical Information and Modeling, 2024 (64); 6778-6798.
DOI: https://doi.org/10.1021/acs.jcim.4c00903

